
인간 장내 마이크로비옴 연구: 개념과 전략
Abstract
The human gut is the natural habitat for a diverse and dynamic microbial ecosystem, and is the primary site of interaction between the host immune system and microorganisms, both symbiotic and pathogenic. Over the years, global leading groups including HMP, Meta-HIT, and IHMC, have investigated the relationship between human and the gut microbiome. The results let us know that the gut microbiota contribute to host physiology including energy harvest from food and development of immune system, and changes in the gut microbiome are associated with many disease phenotypes. Applying suitable model organisms to verify the causal effect of microbial alteration on host’s biological function has played a vital role in aspect of providing various experimental settings that are not performable in human studies. Next generation sequencing platforms and bioinformatics tools have co-evolved, and resulted in an explosion and development of the microbiome studies. In this report, brief overview of the gut microbiome research along with cutting-edge technologies is reviewed in order to help the readers interested in this emerging field.
Keywords:
Intestinal microbiome, Metagenomics, Next generation sequencing, Probiotics인간 마이크로비옴 연구 동향
고세균, 박테리아, 바이러스를 비롯하여 효모 및 곰팡이 등의 진핵생물에 이르기까지, 인체 내·외부에서 미생물은 다양한 생태학적 지위(ecological niche)를 획득하여 인간과 공생 관계를 유지하고 있다. 인체 중에 존재하는 미생물의 수는 인간의 전체 세포 수의 10배 이상이며, 미생물이 보유하는 유전자의 수 역시 인간 유전체의 수백 배 존재한다[1]. 이들 미생물은 인체 내에서 영양소의 흡수와 대사, 면역계와 신경계의 성숙과 발달, 다양한 질환의 발생과 예방에 영향을 미치는 등, 인체와의 상호작용을 통해 주요한 기능을 수행함이 알려져 왔다. 이처럼 인간은 인체 세포와 유전체 외에도 공생하고 있는 수많은 미생물의 세포와 유전자 특성을 섞어 놓은 초유기체(super-organism)로서[1], 인체에 자연적으로 존재하는 모든 미생물 군의 집합체를 인간 마이크로비옴(human microbiome)이라 한다.
인체 미생물(human microbiota)은 피부, 구강, 치아, 생식기, 호흡기, 위장관 등 여러 신체 부위에 존재하나, 이 중 가장 많고 다양한 종류의 미생물을 보유하고 있는 곳은 위장관(gastro-intestinal tract)이다[2]. 위장관은 ~500-1000 개의 다른 종으로 구성된 미생물 군집에 안정된 거주지를 제공하며 미생물-미생물 및 인간-미생물간의 복잡한 상호관계가 이루어지는 주요 신체부위이다[3]. 위장관 내에 존재하는 미생물의 무게는 대략 05-1.5 kg에 이르는 것으로 알려져 있으며, 이들 미생물들의 숙주 항상성 유지, 생물학적·대사적 기능의 중요성과 관련하여 위장관 미생물 군집을 잊혀진 장기(Forgotten Organ)로 일컫기도 한다. 세균의 경우 Firmicutes(35-80%), Bacteroidetes(17-60%), Actinobacteria, Proteo-bacteria, Verrumicrobia 등의 phyla가 위장관에 주로 존재하며, 세균만이 아니라 박테리오파지를 포함한 여러 종류의 바이러스 역시 위장관의 미생물 군집에 중요한 영향을 미친다. 위장관 내 미생물 (특히, 박테리아) 군집의 농도 및 다양성은 위치 별로 상당한 차이가 있는데, 위(stomach) 내의 경우 103~104 정도로 가장 적은 분포를 보이는 반면, 대장(large intestine)의 경우 1 g 당 1011 정도로 가장 많은 미생물이 분포한다[4].
인간 마이크로비옴은 개인, 가족, 인종, 성별, 나이, 식습관, 지역, 생활 방식 등 다양한 원인에 의해 역동적으로 변화한다. 그러나, 각 개인의 미생물 군집을 구성하는 미생물 종(species)의 다양성과는 달리 마이크로비옴을 이루는 주요 기능성 유전자들(core microbiome)은 개체 특성과 환경 조건의 차이에도 상당 부분 공유됨이 밝혀졌다. 인간 마이크로비옴의 기본 구조와 변화 양상, 다양성 및 안정성(stability)에 관한 이해는 전세계적 마이크로비옴 컨소시엄에 의해 주도된 대규모 연구의 역할이 크다. 이 중 대표적인 것들로 미국국립보건원(US National Institutes of Health, NIH)에서 지원을 받는 HMP(Human Microbiome Project), 유럽 연합 집행 기관(European Commission)의 지원을 받는 Meta-HIT (Metagenomics of the human intestinal tract), 인간 마이크로비옴 연구를 통한 데이터 자원 공유를 목적으로 구성 된 IHMC(International Human Microbiome Consortium) 등을 들 수 있다. 특히, Meta-HIT과 HMP의 경우 인체 각 부위의 마이크로비옴의 구조에 대한 기본 자료(reference microbiome)를 제공하고, 미생물 군집이 인간의 건강과 질병에 미치는 직·간접적 영향에 관한 주요 연구 결과를 보고하였다.
그러나, 장내 미생물 군집과 그들의 기능에 대한 연결 고리는 매우 복잡하여, 원인과 결과를 증명할 수 있는 연구 방법론의 모색이 주요 과제로 대두되었다. 오징어, 초파리, C. elegans, 제브라피쉬, 마우스, 돼지 등의 동물 모델은 인간을 대상으로 수행할 수 없는 실험 조건을 적용할 수 있어 호스트와 장내 미생물 간의 상호관계 연구에 널리 이용되어 왔으며, 특히 무균 동물(germ-free animal)의 개발은 미생물과 인체 기능의 인과 관계 이해에 중요한 역할을 하였다[5]. 또한, 장내 미생물 군집이 형성되는 생애 초기의 노출 환경이 유사하고 유전적 요인의 영향력을 평가하는데 유리한 불일치 쌍둥이(discordant twins) 역시 효과적인 연구 모델의 하나로 사용된다[6]. 연구 모델의 선정과 새로운 방법론의 모색은 장내 마이크로바이옴 연구에 필수적이며, 앞으로도 질병과 장내 미생물의 관계를 파악할 수 있는 다양한 질환 모델의 개발이 필요하다.
대용량의 시퀀싱 기술의 발달은 배양 조건의 제약을 벗어나 다양한 환경 중에 존재하는 미생물 군집을 연구하는데 혁신적인 변화와 발전을 가져왔다[7]. 차세대 염기서열분석 (NGS, next generation sequencing) 기술은 배양은 물론 클로닝과 같은 복잡한 단계를 거치지 않으며 기존 연구에 비해 상대적으로 비용 대비 고효율의 연구를 할 수 있는 장점이 있다. 이렇게 얻어진 대용량의 미생물 유전체 자료의 분석을 위해서는 생물정보학(bioinformatics) 기술이 바탕이 되어야 한다.
최근에는 염기서열분석에 기반한 상관 관계 분석에 머무르지 않고, 밝혀진 주요 기능성 미생물 군의 생리적 유용성을 극대화하고 관련 질병 예방에 활용하기 위해, HTS(High-Throughput Screening) 시스템을 이용한 유용 균주의 동정과 분리, culturomics 기법의 도입, 인체 생리활성 검증을 위한 중개연구 등 다양한 전략적 모색이 이루어지고 있다.
장내 마이크로비옴의 정의와 분류
인간의 장내 미생물은 태어날 때부터 유전, 식습관, 생활 습관 등에 따라 개인별로 다양한 군집 구조를 갖는다[9]. 이들 미생물 군집이 지니는 유전자 집합체를 장내 마이크로비옴(intestinal microbiome)으로 정의한다. NGS를 통해 16S rRNA 유전자 분석에 기반한 장내 미생물 군집 구조를 살펴보면, 장내에는 대략 8개의 박테리아 문(phylum)이 존재하며 가장 많은 것은 Firmicutes와 Bacteroidetes이다[11].
장내 미생물 군집의 구조-기능 간의 관계를 이해하는데 유용한 개념 중의 하나로, ‘장유형(enterotypes)’ 을 들 수 있다. Burcelin 등은 39명의 건강한 성인을 대상으로 계통적, 기능적 메타게놈 분석을 수행한 결과, Bacteroides, Prevotella, 그리고 Ruminococcus (또는 기타 Firmicutes genera)에 해당하는 세 가지 뚜렷한 클러스터의 형태로 개인의 장내미생물 구조를 분류할 수 있음을 보고하였다[45]. 모든 연구들에서 뚜렷한 세 개의 장유형의 구분이 발견되는 것은 아니나, 오랜 식습관과 장유형이 상당한 관련이 있으며 Bacteroides 유형은 단백질과 포화지방의 섭취, Prevotella 유형은 탄수화물 및 당류의 섭취 증가와 관련이 있다고 여겨진다[46].
장유형에 따른 분류는 상당히 유용하다. 그러나, 장내 환경은 음식 섭취, 생활 방식, 위생 상태, 항생제를 비롯한 약물 사용 등 환경에 따라 장내 미생물의 군집 구조와 마이크로비옴의 급격한 변화가 수반되는 역동적인 생태 환경으로, 장내 미생물의 복잡한 기능을 이해하기 위해서는 마이크로비옴의 구조적 특성에 관한 보다 깊은 이해가 필요하다.
식습관이 마이크로비옴에 미치는 영향
장기적 식습관의 장내 미생물의 군집에 미치는 영향의 예로, 식물성 탄수화물을 다량 섭취하는 아프리카 지방 마을의 아이들은 이탈리아 도시의 아이들에 비해 Firmicutes 비율이 낮고 Bacteroidetes(특히, Prevotella 와 Xylanibacter)의 비율이 높았다[21]. Prevotella와 Xylanibacter는 셀룰로오즈(cellulose)와 자일란(xylans)의 분해능이 높으며, 짧은 사슬 지방산(short-chain fatty acids; SCFAs)을 증가시킨다. 아프리카 지방 마을 아이들의 장내 미생물 군집은 섬유소가 풍부한 식습관으로부터 최대한의 에너지를 추출해 내도록 적응된 것으로 여겨진다.
섭취된 탄수화물의 형태 역시 장내 미생물 군집과 숙주 생리의 변화를 유도한다. 프리바이오틱스(prebiotics)인 이눌린 (inulin) 섭취 시, Bifidobacterium sp.와 Faecalibacterium prausnitzii가 증가되며[22], 장내 Bifidobacterium sp.를 선택적으로 증가시킨 비만쥐는 고지방식이를 한 비만쥐와 비교해 혈중 LPS (lipopolysaccharides)와 같은 염증성 지표가 감소되었다[23].
장내 미생물의 군집은 고지방식이에 의해서도 급격하게 변화한다. 고지방식이를 먹인 실험쥐의 경우 전체적으로 Bacteroidetes의 감소, Firmicutes와 Proteobacteria의 증가가 관찰되며 군집 구조 자체에도 매우 큰 변화가 유도된다[24]. 한편, 무균쥐에 고지방식이를 한 쥐의 장내 미생물을 먹이면 지방과다증이 정상쥐의 장내 미생물을 먹였을 때보다 더 증가한다. 이는 무균쥐에 이식된 고지방식이 쥐의 장내 미생물 군집으로 비만을 유도할 수 있음을 보여주는 예로 널리 알려졌으나, 아직까지 자세한 기작이 밝혀지지는 않았다[25].
장내 마이크로비옴의 기능
장내 미생물은 인간이 소화하기 어려운 음식들을 소화 가능한 형태로 분해, 전환시켜 에너지의 확보와 소화를 돕는 등의 대사 기능 조절 작용 외에도, 점막 면역계의 성숙과 발달, 신경계와의 상호작용, 인체 항상성 유지와 관련된 다양한 기능을 수행한다[5,12,13].
에너지의 확보
탄수화물은 인간과 장내 미생물에게 중요한 에너지원이다. 하지만 인간이 가지고 있는 효소는 대부분의 탄수화물과 복잡한 형태의 식물성 다당류를 분해 할 수 없다. 인간의 장내 환경, 특히 대장은 1011 cfu/g에 이르는 혐기성 미생물이 사는 생물반응기로, 식물 유래의 펙틴, 셀룰로오즈, 헤미셀룰로오즈 그리고 효소 저항성 전분(resistant starch) 등 인간 스스로 소화하기 어려운 다당류는 미생물에 의해 분해된다[11]. 이 과정에서 acetate, propionate, butyrate와 같은 SCFA들이 생성되며, 이들 산물은 장 상피세포의 에너지원, 면역계의 강화, 염증 저감화, 대사 조절 등 건강에 유익한 영향을 준다.
한편, 이 과정은 장내 미생물 구성원 간의 상호작용에 의해서도 영향을 받는다. 장내 미생물은 서로 영향을 주고 받으며 섭취된 식품의 대사 과정을 복잡하게 변형시키고, 결과적으로 숙주의 대사능과 질병 관련 인자를 변화시킨다. 예로, 장내 고세균의 하나인 Methanobrevibacter smithii와 식물성 다당류의 분해능이 높은 장내세균인 Bacteroides thetaiotaomicron를 무균쥐에 함께 정착시킨 gnotobiotic 모델에서 fructans을 섭취시켰을 때, B. thetaiotaomicron은 더 많은 acetate와 formate를 생성하고 M. smithii가 생성된 formate를 사용하여 메탄을 생산함으로써, 결국 섭취된 비소화성 탄수화물의 발효율 향상 및 에너지 흡수의 증가를 야기한다. 이는 식품 섭취에 따른 에너지 흡수율을 평가할 때, 숙주의 에너지 대사 경로뿐 아니라 개인이 보유하고 있는 미생물 군의 차이와 그로 인한 발효능 및 식품 흡수율의 변화를 함께 고려해야 함을 시사한다.
섭취된 식품을 바탕으로 에너지 확보를 하는 장내 미생물의 역할은 실험쥐를 통해 다양하게 증명되고 있으나, 인간 장내에서의 연구는 간접적으로 진행된다. 예로, 많은 양의 SCFA를 생산하는 비만인 사람은 정상인에 비해 호흡에서 에탄올 수준이 더 높은 것이 확인 되었고[19,20] 이는 미생물에 의해 더 많은 에너지가 확보 되었다는 것을 의미한다.
방어 기능
장내 정상균총(normal flora)은 숙주의 대사과정을 효율적으로 활용하며 안정적인 군집을 형성함과 동시에, 인간과 공생하면서 외부의 위협을 차단하는 기능도 함께 수행한다[29]. 외부에서 유입된 병원균은 장내 상피 세포에 부착하기 위해서 우선 정상균총과 경쟁하게 된다. 무균 동물의 경우 외부 감염원에 매우 민감한데[26], 이는 정상균총이 병원균의 침입과 장내 방어벽(intestinal barrier) 기능의 강화에 중요함을 보여주는 예이다[28]. 정상균총은 특히 장내 점막면역계의 발달과 성숙에 필수 요소로서, 특정 미생물군은 특정 종류의 면역 세포의 분화와 활성화를 유도하여 면역 관용(immune tolerance)과 면역 자극(immune stimulation) 간의 균형을 조절한다. 또한, 정상균총은 박테리오신을 비롯한 항미생물 물질을 생성함으로써 외부에서 침입한 병원균을 억제하기도 한다[30].
각 개인별로 정상균총은 유전, 환경, 식습관과 같은 여러 요인에 따라 그 구성이 다양하지만, 개인 내에서는 그 종류와 수의 균형을 이루며 군집 구조와 안정성을 유지하고 있다. 그러나, 군집의 안정성을 무너뜨리는 외부적 요인들은 정상균총의 방어기능을 약화시켜 결과적으로 감염성 및 염증성 질환의 발생에 영향을 미친다. 대표적인 예로, 항생제의 장기적 복용은 정상적인 장내 미생물 군집의 균형을 무너뜨리고, Clostridium difficile과 같은 특정 균의 과성장을 유도하여 장내 염증 반응을 증가시킨다[27].
나아가, 장내 방어벽 기능이 약화되고 장관점막의 투과성이 증가하면 장관 내에 있던 병원균 외에도 이들의 부산물, 독소, 항원 등이 혈류로 유입되어 면역체계를 자극함으로써, 각종 급·만성 감염성 질환과 자가면역질환 등의 발병에 영향을 주기도 한다.
프로바이오틱스와 프리바이오틱스
AO/WHO 의 정의에 따르면 프로바이오틱스(probiotics)는 ‘적정량을 섭취하였을 때 숙주에 유익한 영향을 미치는 살아있는 미생물’을 지칭한다. 20세기 Elie Metchnikoff에 의해 처음 그 개념이 도입된 이래, ‘장 건강에 도움을 주는 유익한 미생물’의 존재는 주로 lactobacilli 및 bifidobacteria와 같은 유산균을 중심으로 그 효능이 널리 연구되어 왔으며, 오늘날 다양한 프로바이오틱 기능성 제품들에 응용되었다.
프로바이오틱 균주의 효능 중 대표적인 것으로는 항생제 부작용의 감소, 감염성 설사의 치료, 장내 미생물 균형 조절(정장작용), 아토피 증상의 완화, 유당 불내증 증상 완화, 염증성 장질환 증상 완화, 혈중 콜레스테롤 감소 등 장관계 질환 및 각종 대사 질환의 예방 및 증상 완화와 연관된 것으로 보고되었다. 그러나, 프로바이오틱스의 섭취에 의한 장내 미생물 구조 및 숙주의 대사체계, 점막면역체계의 변화는 외부에서 투입된 균들의 성공적인 정착을 전제로 한다. 인간을 대상으로 한 실험들에서 프로바이오틱스 균주의 정착은 주로 단시간만 가능하여, 일반적으로 프로바이오틱 제품은 꾸준히 섭취해야 그 효능을 기대할 수 있다.
프리바이오틱스(prebiotics)는 일반적으로 동물에 의해서는 소화되지 않으나, 프로바이오틱스의 성장을 선택적으로 촉진할 수 있는 물질들로서, inulin, oligofructose, galactooligosaccharides, lactulose 등이 대표적으로 이에 해당한다. 이들 중 일부는 특히 bifidobacteria와 lactobacilli의 선택적 성장을 촉진하여 장내 미생물 구조를 변화시키고, 숙주의 대사 체계 및 면역계의 강화에 기여한다고 알려져 있다.
최근 NGS 기법의 발달로 장내 마이크로비옴 구조 분석이 널리 이뤄진 결과, 새로운 프로바이오틱스와 프리바이오틱스의 발굴이 더욱 가속화되었다. 과거 이들 개념은 유산균과 이들의 생장을 촉진하는 수 종의 난소화성 다당류를 주로 지칭하는데 사용되었다. 그러나, Akkermansia muciniphila, Faecalibacterium prausnitzii, Roseburia intestinalis 등 질병의 예방과 증상 완화와 관련된 기능을 수행하는 다양한 장내 미생물의 역할이 밝혀짐으로써, 이들 유용 미생물들을 프로바이오틱 균주로서 활용하기 위한 많은 노력과 연구가 이뤄지고 있다.
장내 마이크로비옴과 질병
비만, 당뇨 및 대사 증후군
장내 미생물의 비만, 심장질환, 제2형 당뇨 및 대사 증후군과의 관련성은 이들을 예방하기 위한 목적의 마이크로비옴 기반 치료제들이 등장하는 등 마이크로비옴 분야에서 가장 널리 연구되어 온 주제 중 하나이다.
장내 미생물 군집 조성은 숙주의 체중이 변화함에 따라 변화한다. 유전적으로 변형 된 ob/ob 실험용 쥐는 과식 유도 호르몬인 렙틴(leptin) 생성에 유전적 장애가 나타나 자연적으로 비만이 유도된다. 이 실험용 쥐의 맹장(cecum)의 장내 세균 군집을 분석하면, 정상인 일반 쥐에 비해 Firmicutes가 많고 Bacteroidetes가 적게 나타났다[14]. 이와 유사한 변화가 비만인 사람의 분변 미생물에서도 보였으며, 식이 요법을 통해 체중이 감소했을 때 Bacteroidetes가 증가하는 경향을 나타냈다[15]. Bacteroidetes가 식품 섭취 시 칼로리 섭취에 반응 한다는 것을 보여준다. 위장관 우회술(gastric bypass)은 고도비만인 환자를 대상으로 체중 감소와 당뇨, 심장질환의 위험도 감소를 목적으로 시행된다[16]. 연구에 따르면 위장관 우회술의 실시 전·후에도 분변 미생물의 구성이 변화하였는데, 정상 체중군과 비만군과 비교하여 우회술 실시군에서 특히 Gammaproteo-bacteria의 유의적 증가가 나타났다[17]. 정확한 원인은 아직 밝혀지지 않았으나 위장관 우회술의 실시와 그에 따른 식품 섭취 및 소화과정의 변화와 관련이 있다고 여겨진다.
염증성 장질환(IBD, inflammatory bowel disease)
염증성 장질환은 Crohn’s disease 과 ulcerative colitis 를 통합하여 지칭하며, 발병 과정에서 장내 미생물과 조성이 주요 인자로 작용함이 알려져 있는 만성 염증 질환이다. IBD와 장내 마이크로비옴의 연관성은 in vitro, in vivo 실험, 동물 모델 및 인간을 대상으로 한 중개·관찰·실험 연구 등을 포함해 오랜 기간 널리 행해져 왔다. IBD가 진행되면, 장내 미생물 군집의 다양성의 감소와 함께 Proteobacterial Enterobacteriaceae의 증가와 Fecalibacterium prauzsnitzii를 포함한 Firmicutes의 감소가 관찰된다[47]. 그러나, IBD의 발병기작(pathogenesis)에서 장내 미생물이 미치는 인과성(causality)에 관한 연구는 여전히 진행 중이다. 이들 미생물의 불균형이 유전적으로 민감한 숙주(susceptible host)에서 IBD의 발병에 기여하는 것인지, 급성 염증으로 인한 장내 환경의 변화에 따른 결과인지는 추가적인 연구가 필요하다.
자가 면역 질환(autoimmune disease) 및 기타
장내 미생물과 질환과의 관련성은 대사성 질환 및 장관련 질환을 넘어 다양한 신체 부위에서 발생하는 여러 질환에 영향을 미침이 보고되고 있다. 특히, 제1형 당뇨(type 1 diabetes), coeliac disease, Rheumatoid arthritis와 같은 자가면역 질환이 상관관계가 있음이 알려졌다[48]. 한편, 위장관(gastrointestinal tract)과 중추신경계(central nervous system)가 gut-brain axis를 통해 상호작용하고 있으며, ASD(autism spectrum disorder), multiple sclerosis, Alzheimer's disease 등과 같은 neurological disorders의 발생과도 연관됨이 보고되고 있다[49].
장내 마이크로비옴 연구를 위한 기법
동물 모델
장내 미생물과 호스트의 상호관계를 연구하기 위한 다양한 동물 모델이 사용되어 왔다. 각각의 모델은 특정한 수와 종류로 구성된 특이적인 미생물 군집 구조를 가지며, 연구 목적에 따라 적합한 모델을 선택할 수 있다. 예로, 하와이안 갑오징어 (Hawaiian bobtail squid)[31] 는 Vibrio sp. 단일로 이루어진 미생물 군집을 가져 미생물의 유전적 변형과 그 영향을 살펴보는데 용이하고, 미생물-숙주 상호관계 연구에 사용되는 가장 단순한 모델이다. 초파리[32]의 미생물총은 30종 미만으로 구성되어 있고, 혐기성 장내 미생물 배양이 가능하며 숙주를 유전적으로 조작하기 쉬운 장점이 있다. 제브라피시 (zebrafish)[33]는 척추동물과 비슷한 생리학적, 면역학적 특징 및 상당히 복잡한 장내 미생물 군집 구조를 가지고 있으며, 다량의 개체를 대상으로 한 연구에 유용하다. 마우스[34]는 숙주의 유전적 다양성과 장내 미생물의 분류학적 측면에서, 돼지와 더불어 인간과 가장 유사한 모델이다. 인간과는 다르게 무균 또는 유전적으로 조절이 가능하기 때문에 매우 유용하고 가장 널리 이용된다. 장내미생물과 숙주의 복잡한 상관관계 특히 특정 미생물의 인과적 영향을 이해하기 위해서는 앞으로도 다양한 실험적 디자인에 기반한 동물 모델의 개발이 큰 도움이 될 것이다[6].
NGS 및 생명정보학 분석 방법
1970년부터 혐기성 배양을 통해 400-500 개 이상의 세균을 장내에서 분리해왔다. 하지만 배양 방법은 80% 이상의 장내 미생물을 배양 할 수 없다는 단점을 가지고 있다[35]. 1977년 Woese와 Fox는 보존적인 시퀀스와 가변적인 시퀀스가 공존함으로서 박테리아 계통학적 특징을 반영하는 16S rRNA 분자를 발견 하였다[36]. 이후 30여 년 간, 16S rRNA는 TRFLP(terminal restriction fragment length polymorphism), DGGE(denaturing gradient gel electrophoresis), FISH(fluorescence in situ hybridization) 등을 포함한 장내 미생물 군집 구조를 파악에 핵심적인 역할을 하였다[37].
인간 유전체 프로젝트(human genome project) 이후 차세대 염기서열 분석(next generation sequencing; NGS) 기술이 두각을 나타냈으며 심도(depth)와 속도가 증가된 대용량의 유전자 서열 시퀀싱을 상대적으로 낮은 비용으로 실시할 수 있게 되었다. 대표적인 platform으로는 Roche의 454 pyrosequencing과 Illumina의 Hi-seq 2000 등이 있으며, read length와 throughput이 증가 된 장비들이 지속적으로 개발되고 있다.
16S rRNA는 미생물 군집 구조를 파악하는데 획기적으로 기여한 분자임에 틀림없으나, 미생물 유전체의 기능적 측면과 생리학적 중요성에 대한 정보는 가지고 있지 않다[38]. 마이크로비옴의 기능적 측면을 파악하기 위해서는 전체 미생물의 유전체를 시퀀싱하고, 이전에 규명된 유전자와 비교하여 미생물 군집의 기능적인 측면을 연구하는 전체 유전자 염기서열 분석법(whole genome sequencing; WGS)이 필요하다. WGS을 통해 복잡한 군집 안에 희소한 박테리아의 규명을 할 수 있을 뿐만 아니라 대사 또는 생물학적 기능에 대한 미생물 유전자 규명이 가능하다[39]. 그러나, WGS에는 많은 양의 DNA가 필요하며 숙주 DNA에 미생물의 유전체가 오염 될 우려가 있고, 아직까지 많은 유전자들의 기능적인 정보가 아직 규명 되지 있지 않았다는 단점이 있다. 16S rRNA 유전자와 taxonomy 정보에 기반한 마이크로비옴 구조분석(community profiling)과 WGS 을 통한 유전체의 기능성 유전자 분석과정은 Figure 1 에 제시되어 있다.
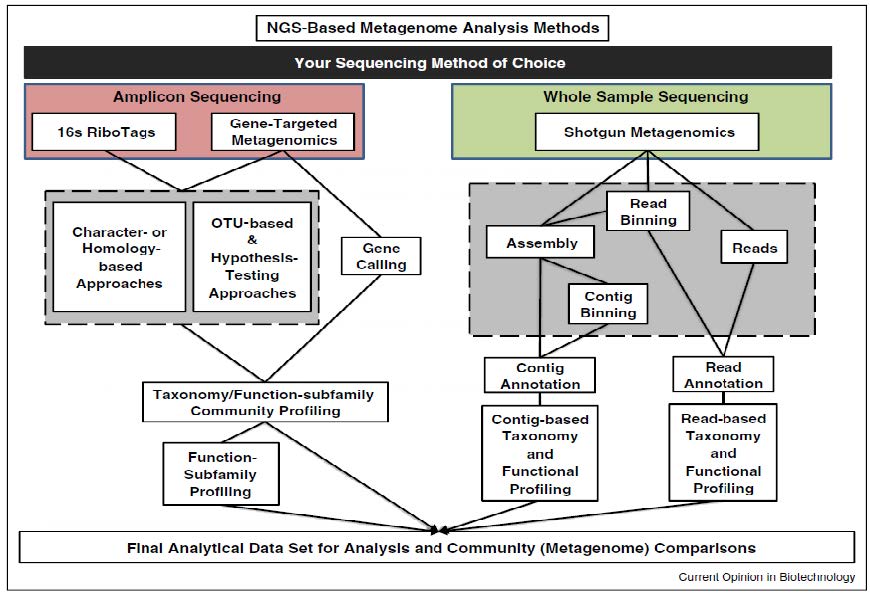
Analysis of metagenomic data from amplicon sequencing or whole-sample shotgun metagenome sequencing (Source: Scholz et al., 2012. Curr Opin Biotechnol [44])
NGS 기법이 발달로 대용량의 염기서열을 보다 쉽게 획득할 수 있게 된 현재, 이들 데이터를 분석하여 의미 있는 정보로 해석 하는 과정이 더욱 중요해졌다. 생물정보학(bioinformatics)을 기반으로 하는 많은 분석 프로그램들이 개발되었고, 16S rRNA와 WGS 데이터에 따라 분석 방식과 알고리즘은 다른 양상을 보인다. 16S rRNA 에 기반한 community 분석은 대표적으로 QIIME[40] 과 mother[41] pipeline이 군집 비교에 주로 이용된다. 분석 과정은 대용량의 시퀀스를 노이즈 필터링, 정렬 (alignment), 분류를 하여 시퀀스 그룹을 근거로 한 OTUs(operational taxonomic units) 표를 생성한다. 다음 샘플 정보와 비교하여 네트워크 분석과 다양성 분석을 실시한다[42]. 반면 전체 유전자 서열 분석(WGS)은 노이즈 필터링, 조립(assembly), 유전자 예측 (prediction) 등을 통해 기존 유전자 정보 데이터 베이스와 비교하여 미생물 군집의 기능을 분석한다.
일반 연구자들이 이러한 분석을 실시하려면 대용량 시퀀스 데이터를 분석 할 수 있는 컴퓨팅 시설 확보가 필요하다. 최근 클라우드 컴퓨팅은 대용량의 시퀀스를 보관, 분석 할수 있는 원격의 저장공간과 파워풀 한 메모리, CPU를 빌려주는 서버 시스템이다. 메타제노믹스(meta-genomics) 분석에서 널리 사용되는 상업적 시스템으로는 클라우드 바이오리눅스(Cloud Bio-Linux)와 아마존(Amazon Elastic Compute Cloud; http://aws.amazon.com/ec2/)이 있다. 더불어 국립 과학 재단에서 지원하는 무료 컴퓨터 클라우드 시스템으로는 DIAG(Data Intensive Academic Grid, http://diagcomputing.org)가 있다.
최근 마이크로비옴 연구 동향
최근 마이크로비옴 연구는 상관관계와 인과관계를 밝히기 위한 실험적 증명을 넘어, 현재까지의 연구 성과를 질환의 예방과 인간의 건강 증진을 위해 적용하기 위한 시도를 향해 나아가고 있다. 마이크로비옴 신약 기술은 2011년, ‘Science지 선정- 10대 breakthrough 기술’ 및 2014년 ‘세계경제포럼 선정- 미래를 바꿀 10대 떠오르는 기술’로 선정되었다. 이를 위해, 유효기능성 미생물의 순수 분리와 발굴 및 효능 평가, 인체 적용가능성 탐색과 관련된 연구가 활발히 진행 중이다.
Conclusion
인간의 몸은 10배 이상의 많은 미생물과 공생 관계를 이루며 살아가고 있다. 특히, 가장 많은 미생물이 사는 장내는 그들이 영양분 섭취와 체계적인 군집 형성을 하기에 최적의 환경을 제공한다. 장내 미생물은 짧은 사슬 지방산(short-chain fatty acids; SCFAs)을 생성하고 숙주가 지닌 효소만으로는 생성 할 수 없는 영양분을 공급하고, 숙주의 대사 및 면역 체계와 깊은 연관을 지닌다. 현재까지 장내 미생물 군집은 비만, 당뇨를 포함한 대사 증후군, 염증성 장질환, 자가 면역 질환의 발생과 관련됨이 보고되어 왔으나, 이들 질환을 유발하는 장내 미생물이 무엇인지는 아직 불분명한 상태이다. 이는 대상자 개개인의 유전적 특성, 인종, 식습관 등 다양한 요인이 복합적으로 작용하기 때문이며, 장내미생물의 기능과 질환과의 인과성에 관한 연구는 앞으 로의 과제라 할 수 있다. 이를 위해, 변수의 통제가 가능한 적합한 동물 모델을 개발하고, 각 동물 모델의 유전체 특성, 정착 미생물의 종류와 수, 인간 소화기관과의 유사성 및 차이점 등을 고려한 분석과 평가기술에 관한 연구가 필요하다. 시퀀싱 비용의 절감에 발맞춘 보다 개선된 생물정보학 분석 도구의 개발 역시 장내 미생물 군집의 구성과 기능에 관한 편차 없는 분석 결과를 도출하는데 선행되어야 할 조건이다. 또한 현재까지의 장내 마이크로비옴 연구는 메타제노믹스(metagenomics)를 위주로 진행되어 왔으나, 특정 조건에서 발현되는 미생물 유전자와 단밸질을 평가하기 위한 메타전사체(meta-transcriptomics), 메타단백질체(metaproteo-mics), 대사체학(metabolomics)적 접근 역시 함께 이루어진다면 통합적인 이해에 더욱 큰 도움을 줄 것이다.
Acknowledgments
본 연구는 한국연구재단 도약연구 (NRF-2010-0029113) 및 서울대학교 보건연구재단에서 일부 지원을 받아 진행되었습니다.
References
-
Turnbaugh, PJ., Ley, RE., Hamady, M., Fraser-Liggett, CM., Knight, R., and Gordon, JI., The human microbiome project, Nature, (2007), 449, p804-810.
[https://doi.org/10.1038/nature06244]

-
Methé, BA., Nelson, KE., Pop, M., et al , A framework for human microbiome research, Nature, (2012), 486, p215-221.
[https://doi.org/10.1038/nature11209]

-
Zoetendal, EG., Vaughan, EE., de Vos, WM., A microbial world within us, Mol Microbiol, (2006), 59, p1639-1650.
[https://doi.org/10.1111/j.1365-2958.2006.05056.x]

-
Eckburg, PB., Bik, EM., Bernstein, CN., et al , Diversity of the human intestinal microbial flora, Science, (2005), 308, p1635-1638.
[https://doi.org/10.1126/science.1110591]

-
Tremaroli, V., Backhed, F.., Functional interactions between the gut microbiota and host metabolism, Nature, (2012), 489, p242-249.
[https://doi.org/10.1038/nature11552]

-
Kostic, AD., Howitt, MR., Garrett, WS., Exploring host-microbiota interactions in animal models and humans, Genes Dev, (2013), 27, p701-718.
[https://doi.org/10.1101/gad.212522.112]

-
Turnbaugh, PJ., Hamady, M., Yatsunenko, T., et al , A core gut microbiome in obese and lean twins, Nature, (2009), 457, p480-484.
[https://doi.org/10.1038/nature07540]

-
Claesson, MJ., Wang, Q., O'Sullivan, O., et al , Comparison of two next-generation sequencing technologies for resolving highly complex microbiota composition using tandem variable 16S rRNA gene regions, Nucleic Acids Res, (2010), 38, pe200.
[https://doi.org/10.1093/nar/gkq873]

-
Spor, A., Koren, O., Ley, R., Unravelling the effects of the environment and host genotype on the gut microbiome, Nat Rev Microbiol, (2011), 9, p279-290.
[https://doi.org/10.1038/nrmicro2540]

-
Turnbaugh, PJ., Ley, RE., Mahowald, MA., Magrini, V., Mardis, ER., Gordon, JI., An obesity-associated gut microbiome with increased capacity for energy harvest, Nature, (2006), 444, p1027-1031.
[https://doi.org/10.1038/nature05414]

-
Backhed, F., Ley, RE., Sonnenburg, JL., Peterson, DA., Gordon, JI., Host-bacterial mutualism in the human intestine, Science, (2005), 307, p1915-1920.
[https://doi.org/10.1126/science.1104816]

-
Round, JL., manian, SK., The gut microbiota shapes intestinal immune responses during health and disease, Nat Rev Immunol, (2009), 9, p313-323.
[https://doi.org/10.1038/nri2515]

- Salonen, A., Palva, A., de Vos, WM., Microbial functionality in the human intestinal tract, Front Biosci, (2009), 14, p3074-3084.
-
Ley, RE., Bäckhed, F., Turnbaugh, P., Lozupone, CA., Knight, RD., Gordon, JI., Obesity alters gut microbial ecology, Proc Natl Acad Sci USA, (2005), 102, p11070-11075.
[https://doi.org/10.1073/pnas.0504978102]

-
Ley, RE., Turnbaugh, PJ., Klein, S., Gordon, JI., Microbial ecology: human gut microbes associated with obesity, Nature, (2006), 444, p1022-1023.
[https://doi.org/10.1038/4441022a]

-
Sjöström, L., Lindroos, A-K., Peltonen, M., et al , Lifestyle, diabetes, and cardiovascular risk factors 10 years after bariatric surgery, J Engl J Med, (2004), 351, p2683-2693.
[https://doi.org/10.1056/NEJMoa035622]

-
Zhang, H., DiBaise, JK., Zuccolo, A., et al , Human gut microbiota in obesity and after gastric bypass, Proc Natl Acad Sci USA, (2009), 106, p2365-2370.
[https://doi.org/10.1073/pnas.0812600106]

-
Samuel, BS., Gordon, JI., A humanized gnotobiotic mouse model of host–archaeal–bacterial mutualism, Proc Natl Acad Sci USA, (2006), 103, p10011-10016.
[https://doi.org/10.1073/pnas.0602187103]

-
Nair, S., Cope, K., Terence, RH., Diehl, AM., Obesity and female gender increase breath ethanol concentration: potential implications for the pathogenesis of nonalcoholic steatohepatitis, Am J Gastroenterol, (2001), 96, p1200-1204.
[https://doi.org/10.1111/j.1572-0241.2001.03702.x]

-
Schwiertz, A., Taras, D., Schäfer, K., et al , Microbiota and SCFA in lean and overweight healthy subjects, Obesity, (2010), 18, p190-195.
[https://doi.org/10.1038/oby.2009.167]

-
Turnbaugh, PJ., Ridaura, VK., Faith, JJ., Rey, FE., Knight, R., Gordon, JI., The effect of diet on the human gut microbiome: a metagenomic analysis in humanized gnotobiotic mice, Sci Transl Med, (2009), 1, p6ra14.
[https://doi.org/10.1126/scitranslmed.3000322]

-
Ramirez-Farias, C., Slezak, K., Fuller, Z., Duncan, A., Holtrop, G., Louis, P., Effect of inulin on the human gut microbiota: stimulation of Bifidobacterium adolescentis and Faecalibacterium prausnitzii, BJN, (2009), 101, p541-550.
[https://doi.org/10.1017/S0007114508019880]

-
Cani, PD., Neyrinck, A., Fava, F., et al , Selective increases of bifidobacteria in gut microflora improve high-fat-diet-induced diabetes in mice through a mechanism associated with endotoxaemia, Diabetologia, (2007), 50, p2374-2383.
[https://doi.org/10.1007/s00125-007-0791-0]

-
Hildebrandt, MA., Hoffmann, C., Sherrill–Mix, SA., et al , High-fat diet determines the composition of the murine gut microbiome independently of obesity, Gastroenterology, (2009), 137, p1716-1724, e1712.
[https://doi.org/10.1053/j.gastro.2009.08.042]

-
Turnbaugh, PJ., Bäckhed, F., Fulton, L., Gordon, JI., Diet-induced obesity is linked to marked but reversible alterations in the mouse distal gut microbiome, Cell Host Microbe, (2008), 3, p213-223.
[https://doi.org/10.1016/j.chom.2008.02.015]

-
Taguchi, H., Takahashi, M., Yamaguchi, H., et al , Experimental infection of germ-free mice with hyper-toxigenic enterohaemorrhagic Escherichia coli O157: H7, strain 6, J Med Microbiol, (2002), 51, p336-343.
[https://doi.org/10.1099/0022-1317-51-4-336]

-
Waaij, V., The ecology of the human intestine and its consequences for overgrowth by pathogens such as Clostridium difficile, Annu Rev Microbiol, (1989), 43, p69-87.
[https://doi.org/10.1146/annurev.mi.43.100189.000441]

-
Bernet, M., Brassart, D., Neeser, J., Servin, A., Lactobacillus acidophilus LA 1 binds to cultured human intestinal cell lines and inhibits cell attachment and cell invasion by enterovirulent bacteria, Gut, (1994), 35, p483-489.
[https://doi.org/10.1136/gut.35.4.483]

-
Hooper, LV., Xu, J., Falk, PG., Midtvedt, T., Gordon, JI., A molecular sensor that allows a gut commensal to control its nutrient foundation in a competitive ecosystem, Proc Natl Acad Sci USA, (1999), 96, p9833-9838.
[https://doi.org/10.1073/pnas.96.17.9833]

-
Guarner, F., Malagelada, J-R., Gut flora in health and disease, The Lancet, (2003), 361, p512-519.
[https://doi.org/10.1016/S0140-6736(03)12489-0]

-
McFall-Ngai, MJ., and Ruby, EG., Symbiont recognition and subsequent morphogenesis as early events in an animal-bacterial mutualism, Science (Washington), (1991), 254, p1491-1494.
[https://doi.org/10.1126/science.1962208]

-
Chandler, JA., Lang, JM., Bhatnagar, S., Eisen, JA., Kopp, A., Bacterial communities of diverse Drosophila species: ecological context of a host–microbe model system, PLoS Genet, (2011), 7, pe1002272.
[https://doi.org/10.1371/journal.pgen.1002272]

-
Roeselers, G., Mittge, EK., Stephens, WZ., et al , Evidence for a core gut microbiota in the zebrafish, ISME J, (2011), 5, p1595-1608.
[https://doi.org/10.1038/ismej.2011.38]

-
Brinkman, BM., Hildebrand, F., Kubica, M., et al , Caspase deficiency alters the murine gut microbiome, Cell Death Dis, (2011), 2, pe220.
[https://doi.org/10.1038/cddis.2011.101]

-
Hayashi, H., Sakamoto, M., Benno, Y., Phylogenetic analysis of the human gut microbiota using 16S rDNA clone libraries and strictly anaerobic culturebased methods, Microbiol Immunol, (2001), 46, p535-548.
[https://doi.org/10.1111/j.1348-0421.2002.tb02731.x]

-
Woese, CR., Fox, GE., Phylogenetic structure of the prokaryotic domain: the primary kingdoms, Proc Natl Acad Sci USA, (1977), 74, p5088-5090.
[https://doi.org/10.1073/pnas.74.11.5088]

-
Nocker, A., Burr, M., Camper, AK., Genotypic microbial community profiling: a critical technical review, Microbial Ecol, (2007), 54, p276-289.
[https://doi.org/10.1007/s00248-006-9199-5]

-
DeLong, EF., and Pace, NR., Environmental diversity of bacteria and archaea, Syst Biol, (2001), 50, p470-478.
[https://doi.org/10.1080/106351501750435040]

-
Bik, EM., Eckburg, PB., Gill, SR., et al , Molecular analysis of the bacterial microbiota in the human stomach, Proc Natl Acad Sci USA, (2006), 103, p732-737.
[https://doi.org/10.1073/pnas.0506655103]

-
Caporaso, JG., Kuczynski, J., Stombaugh, J., et al , QIIME allows analysis of high-throughput community sequencing data, Nat Methods, (2010), 7, p335-336.
[https://doi.org/10.1038/nmeth.f.303]

-
Schloss, PD., Westcott, SL., Ryabin, T., et al , Introducing mothur: open-source, platformindependent, community-supported software for describing and comparing microbial communities, Appl Environ Microbiol, (2009), 75, p7537-7541.
[https://doi.org/10.1128/AEM.01541-09]

-
Hamady, M., and Knight, R., Microbial community profiling for human microbiome projects: Tools, techniques, and challenges, Genome Res, (2009), 19, p1141-1152.
[https://doi.org/10.1101/gr.085464.108]

- Elaine, H., Jia, VL., Thanos, A., Hutan, A., Jeremy, KN., Understainding the role of gut microbiome-host metabolic signal disruption in health and disease, Cell, (2011), 19, p349-359.
-
Scholz, MB., Lo, CC., Chain, PS., Next generation sequencing and bioinformatic bottlenecks: the current state of metagenomic data analysis, Curr Opin Biotechnol, (2012), 23, p9-15.
[https://doi.org/10.1016/j.copbio.2011.11.013]

-
Burcelin, R., Serino, M., Chabo, C., et al , Metagenome and metabolism: the tissue microbiota hypothesis, Diabetes Obes Metab, (2013), 15, p61-70.
[https://doi.org/10.1111/dom.12157]

-
Wu, GD., Chen, J., Hoffmann, C., et al , Linking longterm dietary patterns with gut microbial enterotypes, Science, (2011), 334, p105-108.
[https://doi.org/10.1126/science.1208344]

-
Wu, GD., Lewis, JD., Analysis of the Human Gut Microbiome and Association with Disease, Clin Gastroenterol Hepatol, (2013), 11, p774-777.
[https://doi.org/10.1016/j.cgh.2013.03.038]

-
McLean, MH., Dieguez, D. Jr., Miller, LM., et al , Does the microbiota play a role in the pathogenesis of autoimmune diseases?, Gut, (2014).
[https://doi.org/10.1136/gutjnl-2014-308514]

-
Wang, Y., Kasper, LH., The role of microbiome in central nervous system disorders, Brain Behav Immun, (2014), 38, p1-12.
[https://doi.org/10.1016/j.bbi.2013.12.015]
