
유전자 네트워크 분석을 통한 심근증 마커유전자 탐색 연구
Abstract
Cardiomyopathy is a heterogeneous disease with structural and functional abnormalities in the heart muscle, which is characterized by a prognosis of heart failure. Recently, several genes related to this have been found. In this study, we aimed to investigate marker genes that can predict the prognosis of heart failure in cardiomyopathies due to genetic factors through network analysis using microarray data.
GSE1145 data of Gene Expression Omnibus was used as microarray data. 11 of normal, 12 of idiopathic dilated cardiomyopathy, 11 of ischemic cardiomyopathy and 5 of hypertrophic cardiomyopathy were used respectively. The gene network was constructed based on the expression correlation data corresponding to the heart-left ventricle mRNA type of the genotype-tissue expression v5 group, and the centrality analysis was performed using the R program.
In the case of heart failure due to cardiomyopathy, a total of 73 genes were specifically regulated. The network analysis of these genes showed high centrality of 10 genes including C1QTNF7, ECM2 and FAM188A. In the 2-mode network analysis between the above genes and the genes responsible for cardiomyopathy, 26 genes including ACTC1, ACTN2, BAG3 and DES showed a high centrality in DCM. In HCM, 10 genes including ACTC1 and ACTN2 showed a significant high centrality.
Genes with high centrality in 1-mode network analysis are likely to play an important role in the development of cardiac failure as a prognosis for cardiomyopathy and may therefore be a target for research and treatment of heart failure. Genes with high centrality in the 2-mode network analysis may be used as markers to predict heart failure due to myocardial prognosis through routine diagnostic tests.
Keywords:
cardiomyopathy, heart failure, differential expression analysis, network analysis, marker geneIntroduction
심근증(cardiomyopathy)은 관상동맥질환이나 고혈압, 판막증, 선천적 심장병등의 심혈관계 질환 없이 심장근육에 구조적, 기능적 이상이 생기는 이질성 질병군으로, 심장의 심실이 비정상적으로 비대해 지거나 확장이 되는 특징을 보인다[1]. 심근증은 심부전(heart failure)을 동반하기도 하는데 이는 심장기능의 저하로 신체에 혈액을 정상적으로 공급해 주지 못하는 데서 비롯된다[2]. 또한 심근증은 심장돌연사와 심부정맥의 주요한 원인이며 특히 아동기 심장돌연사의 중요한 원인 중 하나이다[3,4].
최근 심근증의 발병과 관련이 있는 여러 유전자들이 발견되고 있으며 심근증의 종류에 따른 특이적인 유전자들이 밝혀졌다. 유전성 HCM환자의 80%는 myosin heavy chain 7 (MYH7) 유전자와 myosin binding protein C (MYBPC3) 유전자의 상염색체 우성 돌연변이를 가지고 있다[5]. MYH7의 변이는 액틴 결합(actin binding)도메인, 아데노신트리포스파타아제(ATPase)도메인, 힘을 전달하는(force transmission)도메인 등 중요 부위의 아미노산을 다른 아미노산으로 치환시켜 심장의 기능이 저하되게 하고, MYBPC3의 변이는 주로 종결 코돈이 조기에 발현(nonsense mutation) 되거나 틀 이동(frame shifting)을 일으켜 온전한 단백질을 형성하지 못하게 하여 단백질이 정상적으로 기능을 못하게 한다[6,7]. 유전성 DCM에서는 Titin (TTN) 유전자의 변이가 많이 나타나는데, 주로 넌센스 변이, 틀 이동 변이, 스플라이싱 변이, 복제수 변이(copy number variation)가 일어나며, 이로 인해 직렬삽입(tandem insertion)이나 단백질 절삭(protein truncation)이 일어나 titin의 구조가 변하게 된다[8]. ARVC는 심근세포간 접합부인 개재판 (intercalated disc)의 데스모좀 단백질의 기능 이상으로 일어나는데, plakophilin-2 (PKP2), desmoglein-2 (DSG2), desmoscollin-2 (DSC2), desmoplakin (DSP), junctional plakoglobin (JUP) 유전자들이 데스모좀 단백질의 변이와 관련이 있는 유전자들이다[9]. RCM에서는 Troponin I Type 3 (TNNI3) 유전자의 변이가 가장 많이 관찰 되는데 TNNI3 유전자는 얇은 섬유 단백질의 하나인 cardiac troponin I (cTnI)를 암호화 하고 있다[10]. LVNC에서는 선천적 심장질환이 동반되지 않는 경우 주로 Z선(Z-line)을 암호화하고 있는 LIM domain-binding protein 3 (LDB3) 유전자 등에서 변이를 보인다[11]. 유전성 심근증과 관련이 있는 유전자들은 Table 1에 요약하였다.
심근증의 주요한 예후 중 하나인 심부전은 심장의 구조적 또는 기능적 이상으로 인해 심장이 혈액을 받아들이는 이완기능이나 혈액을 내보내는 수축기능이 감소하여 신체 조직에 필요한 혈액을 제대로 공급하지 못하여 발생하는 질환군이다[12]. 심부전은 주로 심근질환, 관상동맥질환, 고혈압 및 심근증에 의해서 발병하는데, 특히 심근증과 연관이 있는 유전자 변이는 심부전의 다양한 병리학적 측면에 영향을 미친다[13]. 유전적인 원인에 의한 심부전에서 가장 빨리 나타나는 임상적 특징은 심실 공간의 확장 없이 심실벽이 비후해 지거나, 심실벽이 얇아지거나 두께의 변화 없이 심실의 공간이 확장되는 변화를 보이는 것이다. 각각의 심실 구조 변화는 특이적인 혈류역학적 변화를 가지고 오는데, 심실벽이 비후해지면 심장의 수축 기능에는 이상이 없지만 이완기에 이상이 생기는 반면, 심실이 확장되면 심장의 수축기능이 저하된다. 이러한 임상적 증상이 나타나면 HCM이나 DCM으로 진단이 된다[14]. 전 세계적으로 심부전환자들이 증가함에 따라 이에 대한 이환율과 사망률 그리고 의료비용이 사회적 문제로 대두되고 있다. 미국의 경우에는 연간 50만명이 새로 진단을 받고 37억 달러의 비용이 지불되며 5만명이 사망한다[15]. 한국의 경우에도 마찬가지로 심부전 환자가 빠르게 증가하고 있는데 2002년에 약 0.75%였던 유병률이 2013년에는 약 1.53%로 증가했으며 2040년에는 약 3.35%정도로 증가할 것이라 예측된다[16]. 심부전 환자들의 생존율이 개선 되기는 했지만 아직 5년 생존율은 많은 암과 비슷할 정도로 높은 편이다[17]. 또한 말기 심부전 환자의 경우에는 1년 생존율이 50%정도로 낮을 뿐만 아니라 인공심장이나 심장이식 등 고비용 치료가 필요하기 때문에 심부전에 대한 조기진단의 중요성이 커지고 있다.
본 연구에서는 여러 심근증으로 인한 심부전 환자들의 유전자 발현 데이터를 통해 심부전에서 특이적으로 발현량이 조절되는 유전자들을 선별하여 유전자 네트워크를 구성하고, 이에 대한 소셜 네트워크 분석을 통해 심근증의 예후로 심부전이 나타나게 하는데 중요한 역할을 하는 유전자를 찾고자 하였다. 한편, 심근증의 원인이 되는 유전자들과의 2-mode 네트워크 분석을 통해 순환혈액으로 이루어지는 통상적인 진단검사로 심근증의 예후로 심부전을 예측 할 수 있는 마커유전자를 탐색 하였으며 결과적으로 심근증의 예후로서 심부전의 예측에 활용 가능한 유전자 마커들을 제시할 수 있었다.
Methods
Affymetrix microarray data
본 연구에 사용된 마이크로어레이 데이터는 공용 기능 유전체 데이터베이스인 NCBI의 Gene Expression Omnibus (http://www.ncbi.nlm.nih.gov/geo/, GEO)에서 얻었으며 Series GSE1145 데이터를 사용하였다. GEO GSE1145의 title은 “changes in cardiac transcription profiles brought about by heart failure”로 다양한 심근증으로 인한 심부전 환자 들 및 심장질환이 없는 사람의 심장 조직을 채취해 [HG-U133_Plus_2] Affymetrix Human Genome U133 Plus 2.0 Array로 유전자 발현 분석을 실시 한 결과들이다[18]. 이중에서 실험군으로 12개의 Idiopathic dilated Cardiomyopathy (GSM25801~12), 11개의 Ischemic Cardiomyopathy (ICM) (GSM 18477~87), 5개의 Hypertrophic Cardiomyopathy (GSM25818~22) 데이터를, 대조군으로 11개의 Normal (GSM18442~52) 데이터를 사용하였다. NCBI GEO (https://www.ncbi.nlm.nih.gov/geo/down load/?acc=GDS651)에서 확장자가 ‘.cel’인 형식의 파일을 다운로드하여 분석에 사용하였다.
Expression correlation data
다양한 표현형 및 분자적 데이터와 분석도구를 제공하는 GeneNetwork (www.genenetwork.org, GN)를 이용하여 유전자 네트워크를 구성하였다[19]. GN으로부터 the Genotype-Tissue Expression v5 (GTEx v5) group의 Heart-left Ventricle mRNA type에 해당하는 발현 상관성 데이터를 수집하였다. GTEx project는 유전자 발현이 인체의 여러 기관에서 어떻게 조절되는지를 연구하기 위해 발현 상관성 데이터를 생성한다. Version5 까지는 175개체로부터 43개의 조직을 통해 1641개의 RNA sequencing 데이터를 얻었으며, 연구에 사용된 Heart-left Ventricle mRNA type의 데이터는 246개의 샘플을 통해 생성되었다[20].
Differentially expressed genes (DEGs) and Co-expression genes (Co-EGs) analysis
다운로드한 발현량 데이터는 R 프로그램을 이용하여 데이터 전처리 및 통계분석을 실시하였다. 데이터 전처리는 standard Affymetrix algorithm인 Microarray Suite 5.0 (MAS5)로 수행하였다. 먼저 각 실험 별 비특이적인 반응 혹은 슬라이드의 이물질에 의한 반응을 보정해주기 위해 regional adjustment를 통한 background correction을 진행하였다. 각 실험간 환경이나 기기적 조건에 의해 생길 수 있는 편향을 보정하기 위해 스케일링기법을 통한 normalization을 진행한 뒤 Tukey biweight average 법을 통해 여러 probe의 발현강도를 하나로 맞춰주는 summarization을 수행하였다[21,22]. 전처리가 끝난 데이터의 발현강도를 log2 스케일로 변환시킨 뒤 flag 값을 이 용하여 발현이 잘 안된 유전자의 결과를 제외한 후 실험군과 대조군의 비교를 위해 t-test 분석을 실시하였다. Normal 검체의 결과를 idiopathic dilated cardiomyopathy, ischemic cardiomyopathy, hypertrophic cardiomyopathy 검체의 결과와 각각 비교하여 False Discovery Rate (FDR)가 0.05이하이고 발현량이 2배 이상 차이가 나는, 즉 fold change의 절대값이 1보다 큰(two fold change) 유전자만을 DEGs로 선별하고 각각 Comparison 1,2,3으로 명명하였다. Type 1 error의 보정을 위해 FDR의 %를 다양하게 조절하여 발현량에 유의한 차이를 보이는 유전자의 수를 조절하였 다[23].
본 연구에 사용된 실험군의 검체들은 다양한 심근증 환자들 중에서 심부전의 예후를 보이는 환자들에게서 채취되었기 때문에 실험군에서 공통으로 발현이 조절되는 유전자들은 심근증에서 심부전의 예후가 나타나게 하는 경로에 속해 있는 유전자들이라 할 수 있다. 위의 방법으로 세 개의 DEGs그룹을 얻을 수 있는데, 이 세 그룹에 모두 속하는 유전자들을 찾아 idiopathic dilated cardiomyopathy, ischemic cardiomyopathy, hypertrophic cardiomyopathy에서 공통으로 발현량이 조절되는 Co-EGs를 선별하고 comparison 4로 명명하였다(Table 2)
Functional annotation
DAVID (the Database for Annotation, Visualization and Integrated Discovery)는 웹 기반의 생물학적 해석 시스템으로, 대규모의 유전자 및 단백질 목록에서 생물학적 의미를 체계적으로 추출하기 위한 도구이다[24]. 이를 통해 마이크로어레이 분석을 통해 얻은 결과들을 분석 할 수 있는데, “Functional Annotation Chart” 기능을 통해 Gene Ontology, General Annotation 등의 여러 데이터베이스에 수록된 정보들을 얻을 수 있으며 이에 대한 p-value와 adjusted p-value를 확인 할 수 있다. David를 통해 위의 Co-EGs에 대한 Functional annotation을 수행하였다.
Gene network analysis
유전자 발현 데이터를 통해 구성된 유전자 네트워크의 구조를 연구하기 위해 소셜네트워크 분석법을 이용하였다. 이를 통해 네트워크의 핵심 유전자들과 생물학적 시스템을 조절하는데 중요한 역할을 하는 유전자들을 탐색하였다[25].
본 연구에서는 1-mode 네트워크분석과 2-mode 네트워크 분석을 진행하였으며, R 프로그램의 ‘sna’ package를 이용하여 유전자 네트워크를 구성하고 소셜네트워크 분석을 실시하였다. 1-mode 네트워크 분석을 위해 앞서 선별한 Co-EGs를 노드로, GTEx v5의 Heart-left Ventricle mRNA type의 유전자들간 발현 상관성을 연결선으로 하는 네트워크를 구성하였다. 노드들 간의 interaction 연결은 Pearson correlation coefficient (PCC)의 절대값을 기준으로(≥0.5) 하였다. 2-mode 네트워크는 서로 성질이 다른 대체간의 관계로 구성된 네트워크로, 본 연구에서는 Co-EGs와 DCM 및 HCM의 원인이 되는 유전자들 사이의 관계를 2-mode 네트워크로 구성하였으며, 노드들 간 Pearson correlation coefficient의 절대값(≥0.6)을 기준으로 해당 interaction만 연결선을 형성하였다. 2-mode 네트워크의 행렬은 다음과 같다.
A : 선천적으로 원인이 되는 유전자들을 행, Co-EGs를 열로 지정하여 구성한 행렬
B : 전체 네트워크 행렬((n+m)*(n+m))
n : 행 노드, 즉 선천적으로 원인이 되는 유전자들의 개수
m : 열노드, 즉 Co-EGs의 개수
다음은 위의 방법으로 구성한 네트워크에 대해 소셜네트워크의 중심성 분석을 진행하였다. 분석 요소로는 각 노드가 가지는 연결선의 수인 연결정도 중심성, 한 노드가 다른 노드들과 평균적으로 얼마나 가깝게 위치하는지에 대한 근접중심성, 한 노드가 두 개의 다른 노드의 가장 짧은 경로에 위치하여 노드들의 허브역할을 하는지에 대한 중개중심성이 있다. 근접중심성은 아래와 같이 구할 수 있다.
C(i) : 근접중심성
d(i,j): 두 노드 사이의 거리
n : 노드의 수
어느 한 노드가 다른 노드로부터 도달되어지지 않는 경우 근접중심성은 0이 되므로 이를 보완하기 위해 조화중심성을 이용하였다. 조화중심성은 아래와 같이 구할 수 있는데, 본 연구에서 구성한 네트워크에는 모든 노드들과 연결선을 형성하지 않는 노드들이 존재하므로 근접중심성 대신 조화중심성을 사용하였다. 이후에 나오는 근접중심성은 모두 조화중심성을 뜻한다[26].
C(h) : 조화중심성
d(i,j):: 두 노드 사이의 거리
n : 노드의 수
중개중심성은 아래의 식을 통해 구한다.
C(v) : 중개중심성
σst : 노드 s에서 노드 t까지 최단경로의 수
σst(v): 노드 s에서 노드 t까지 최단경로 중 노드 v를 지나는 경로의 수
산출된 중심성 지표들은 네트워크의 규모에 따른 차이가 존재하므로 모든 수치를 정규화하는 작업을 수행하였다. 중심성 지표의 유형에 따라, 네트워크의 방향성 유무를 고려하여 정규화 작업을 수행하였다.
Results
DEGs and Co-EGs analysis using microarray data
본 연구에서는 다양한 종류의 심근증 환자들 중 심부전의 예후를 보이는 사람들의 심장에서 발현되는 유전자들의 정보를 이용하여 심근증의 종류별로 차별 발현되는 유전자들과 심근증에서 심부전의 예후가 나타나게 하는데 공통으로 발현이 조절되는 유전자들을 선별하였다. Idiopathic dilated cardiomyopathy검체의 결과와 normal검체의 결과를 비교한 comparison1에서는 발현량이 증가하는 유전자가 310개, 감소하는 유전자가 314개, 총 624개의 DEGs가 선별되었다. Ischemic cardiomyopathy검체의 결과와 normal검체의 결과를 비교한 comparison2에서는 발현량이 증가하는 유전자 403개, 감소하는 유전자 161개, 총 564개의 DEGs가 선별되었다. Hypertrophic cardiomyopathy검체의 결과와 normal검체의 결과를 비교한 comparison3에서는 발현량이 증가하는 유전자는 240개, 감소하는 유전자는 123개, 총 363개의 DEGs가 선별되었다. Comparison4에는 발현량이 증가하는 유전자 55개, 감소하는 유전자 18개, 총 73개의 Co-EGs가 선별되었다(Table 3).
Functional annotation
David를 통해 전체 Co-EGs와 up-regulated 및 down-regulated되는 유전자들의 Functional annotation을 수행하였다. 전체 Co-EGs에 대한 주요한 내용은 ‘UP_KEYWORDS’ 카테고리의 Secreted, Signal, Collagen, ‘GOTERM_CC_DIRECT’ 카테고리의 proteinaceous extracellular matrix, extracellular region, extracellular space, collagen trimer, extracellular matrix 등이 있다. Up-regulated되는 유전자들의 주요한 내용은 ‘UP_KEY WORDS’ 카테고리의 Secreted, Extracellular matrix, ‘GOTERM_CC_DIRECT’ 카테고리의 extracellular region, extracellular space, ‘GOTERM_BP_DIRECT’ 카테고리의 extracellular matrix organization, axonogenesis 등이 있다. Down-regulated되는 유전자들의 주요한 내용은 ‘UP_KEYWORDS’ 카테고리의 Actin-binding, Myosin, ‘GOTERM_MF_DIRECT’ 카테고리의 actin filament binding, ‘GOTERM_CC_DIRECT’ 카테고리의 sarcomere, myosin complex등이 있다.
Gene network analysis
1-mode 네트워크분석을 위해 차별 및 공통발현 조절 유전자 분석으로 선별한 Co-EGs를 노드로, GTEx v5 Heart-left Ventricle mRNA type의 PCC절대값 0.5이상의 interaction을 연결선으로 구성한 가중치가 없는 무방향 네트워크와 PCC절대값 0.7이상의 interaction을 연결선으로 구성한 가중치가 없는 무방향 네트워크를 구성하였다(Figure 1).
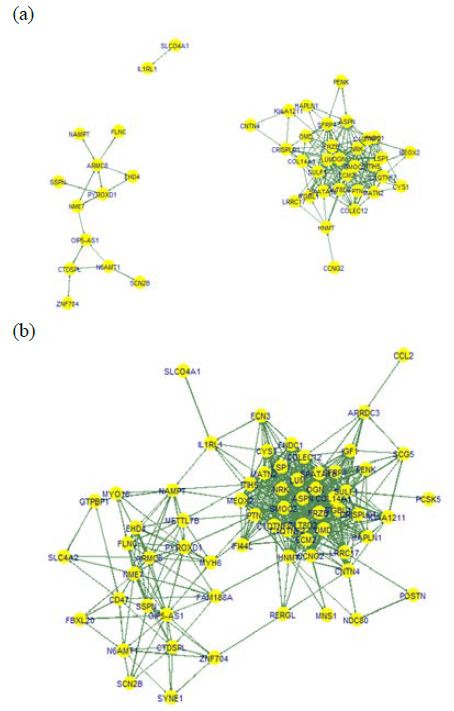
1-mode gene network that composed of Co-EGs. (a) Non-weighted and non-directional network which absolute value of Pearson’s correlation coefficient is above 0.7 (b) Non-weighted and non-directional network which absolute value of Pearson’s correlation coefficient is above 0.5.
PCC절대값 0.7이상의 interaction으로 구성한 네트워크의 전체적인 형상을 보면 크게 두 개의 서브네트워크가 관찰되는데 하나의 네트워크는 up-regulated되는 유전자들로 구성이 되고 다른 하나는 up-regulated되는 유전자들과 down-regulated되는 유전자들로 함께 구성된다. 이는 NME7과 OIP5-AS1 유전자들의 연결선으로 인해 up-regulated되는 부분과 down-regulated되는 부분으로 나뉜다. PCC절대값이 0.5 이상인 interaction을 연결선으로 네트워크를 구성하면 두 서브네트워크 사이에 연결선이 형성된다. PCC절대값이 0.5 이상인 interaction을 연결선으로 구성한 네트워크의 소셜네트워크 중심성 분석을 실시하면, 각 노드에 연결된 연결선의 수를 나타내는 연결정도 중심성은 ECM2가 가장 높게 확인되며 SMOC2, OGN, SULF1, FRZB, LUM의 순서로 높은 결과를 보인다. 하나의 노드에서 각각의 노드 사이의 평균 최단거리의 역수로 나타내어 지는 근접중심성은 SMOC2가 가장 높고 ECM2, SULF1, OGN, NRK, SPATA18의 순서로 높음을 확인하였다. 노드와 노드를 연결해 주는 정도를 나타내는 중개중심성은 HNMT가 가장 높았으며 PTN, NAMPT, FAM188A, ITIH5, MYH6의 순서로 높았다. 위의 중심성 분석에서 연결정도 중심성과 근접중심성은 노드의 수가 많은 up-regulated되는 유전자들로 구성된 서브네트워크에 속해있는 유전자들이 높았으나, 중개중심성은 서브네트워크가 인접한 부분에 위치하여 서브네트워크 간에 연결선을 형성하는데 중요한 역할을 하는 유전자들이 높다는 것을 알 수 있었다.
다음으로 Co-EGs와 DCM 및 HCM의 원인이 되는 유전자들간에 2-mode 네트워크를 구성하여 중심성 분석을 통해 심근증의 예후 중 심부전과 관련이 있는 유전자들을 유추하였다. Co-EGs와 DCM의 원인이 되는 유전자 및 Co-EGs와 HCM의 원인이 되는 유전자를 노드로, GTEx v5 Heart-left Ventricle mRNA type의 PCC절대값 0.6이상의 interaction을 연결선으로 구성한 가중치가 없는 무방향 네트워크를 각각 구성하였다(Figure 2). Co-EGs와 DCM의 원인이 되는 유전자간의 2-mode 네트워크에서 DCM의 원인이 되는 유전자를 중심으로 중심성 분석 결과, 연결정도 중심성은 VCL이 가장 높았으며 DSG2, DSP, ILK, MYPN, NEXN, PKP2의 순서로 높은 것으로 확인되었다. 근접중심성은 VCL이 가장 높았으며 DSG2, DSP, PKP2, MYPN, NEXN, ILK의 순서로 높았다. 중개중심성의 경우 PSEN1이 가장 높았고 ILK, RBM20, VCL, LAMP2, MYPN, FKTN의 순서로 높은 결과를 보였다. 같은 2-mode 네트워크에서 Co-EGs를 중심으로 중심성 분석을 수행하여 연결정도 중심성, 근접중심성, 중개중심성이 높게 산출되는 유전자들을 확인하였다.
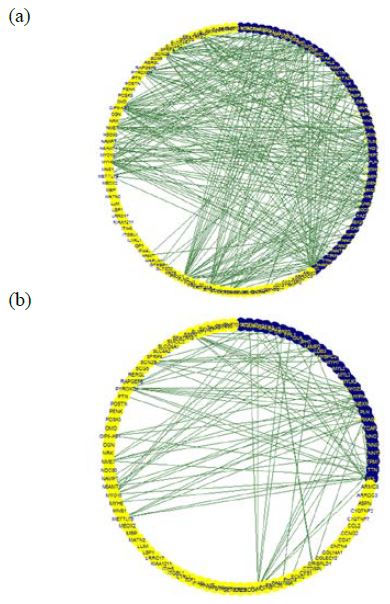
2-mode gene network which absolute value of Pearson’s correlation coefficient is above 0.6. (a) Non-weighted and non-directional that composed of Co-EGs and the genes causing DCM (yellow nodes are Co-EGs and blue nodes are DCM genes). (b) Non-weighted and non-directional that composed of Co-EGs and the genes causing HCM (yellow nodes are Co-EGs and blue nodes are HCM genes).
다음으로 Co-EGs와 HCM의 원인이 되는 유전자간의 2-mode 네트워크에서 HCM의 원인이 되는 유전자를 중심으로 중심성 분석을 하여 세 가지 중심성의 값이 높은 유전자들을 선별할 수 있었다. 마찬가지로, 같은 2-mode 네트워크에서 Co-EGs를 중심으로 중심성 분석을 수행하여 각 데이터셋 별로 높은 중심성을 가지는 것으로 확인되는 유전자들을 결과로 얻을 수 있었다. 1-mode 네트워크와 2-mode 네트워크의 상위10개 유전자의 중심성 분석 결과는 Table 4를 통해 확인 할 수 있다.
이렇게 얻은 중심성의 결과들을 하나의 지표로 통합하기 위해 하나의 항목이 각각의 지표에서 차지하는 비율을 구하여 세가지 지표를 표준화하고, 각 유전자가 가지는 세 지표의 평균 값을 구하였다. 결과적으로 통합적인 중심성의 정도는 1-mode 네트워크에서 HNMT, PTN, ITIH5, SMOC2, SULF1, NAMPT, FAM188A, ECM2, OIP5-AS1, C1QTNF7가 높게 확인되었다. 2-mode 네트워크에서는 DCM의 경우 PSEN1, ILK, VCL, RBM20, MYPN, LAMP2, PKD1, FKTN, DSG2, DSP의 순으로 높은 중심성을 보였고, HCM의 PLN, VCL, MYOZ2, LAMP2, MYPN, PRKAG2, NEXN, TTN의 순으로 중심성이 높게 확인되었다.
Functional protein association networks analysis
Co-EGs와 DCM 및 HCM의 원인이 되는 유전자들간에 존재하는 protein interaction을 알아보기 위해 Search Tool for the Retrieval of Interacting Genes/Proteins (http://string-db.org/, STRING)의 combined score를 바탕으로 2-mode 네트워크를 구성하여 중심성 분석을 실시하였다. ‘Experimental/Biochemical Data’, ‘Co-Expression’, ‘Gene Fusions’ 등 기능적 연관성을 나타내주는 지표들을 통합한 Combined Score가 0.4 이상인 interaction을 기반으로 Co-EGs와 DCM 및 HCM의 원인 유전자들 사이에 2-mode 네트워크가 구성되었다. 이렇게 구성한 단백질 네트워크는 R 프로그램의 ‘sna’ package를 이용하여 연결정도 중심성, 근접중심성, 중개중심성을 척도로 하는 중심성 분석을 한 뒤, 유전자 네트워크 분석 결과와 통합하기 위해 각 중심성 척도에서 각각의 유전자가 차지하는 비율로 유전자의 중심성을 나타내었다.
Co-EGs와 DCM의 원인이 되는 유전자 간의 2-mode 단백질 네트워크에서 DCM의 원인이 되는 유전자를 중심으로 한 중심성 분석 결과, ACTC1, DMD, TCAP, SCN5A, BAG3의 순서로 중심도가 높게 산출되었다. Co-EGs와 HCM의 원인 이 되는 유전자간의 2-mode 단백질 네트워크에서 HCM의 원인이 되는 유전자를 중심으로 중심성 분석을 수행한 결과, ACTC1, ACTN2, TCAP, ANKRD1, LDB3, MYH7, MYOZ2, VCL의 순서로 높은 중심도를 나타내었다.
이렇게 얻은 단백질 네트워크의 중심도 수치를 유전자 네트워크의 중심도 수치에 더하여 protein interaction으로 보정된 유전자 네트워크의 중심도 데이터를 구할 수 있었다.
Gene selection
DCM과 HCM의 원인 유전자들 중, 심근증에서 심부전의 예후를 보이는데 의미가 있는 유전자를 선별하기 위해 로지스틱 회귀분석을 실시하였다. 연속형인 독립변수는 위에서 얻은 보정된 중심성 데이터로 설정하고, 범주형인 종속변수는 유전질환에 대한 데이터 베이스인 Online Mendelian Inheritance in Man (https://www.omim.org/, OMIM)에서 얻는 심부전의 발병 유무로 설정하였다. 여기서 유전자 네트워크와 단백질 네트워크 모두에서 연결선을 형성하고 있지 않은 ABCC9은 분석에서 제외하였다. R 프로그래밍 언어를 통해 로지스틱 회귀분석을 실시하여 DCM과 HCM에서 각각 8.47x10-4, 1.95x10-2의 p-value를 통해 통계적 유의성을 확인하였다(Figure 3).
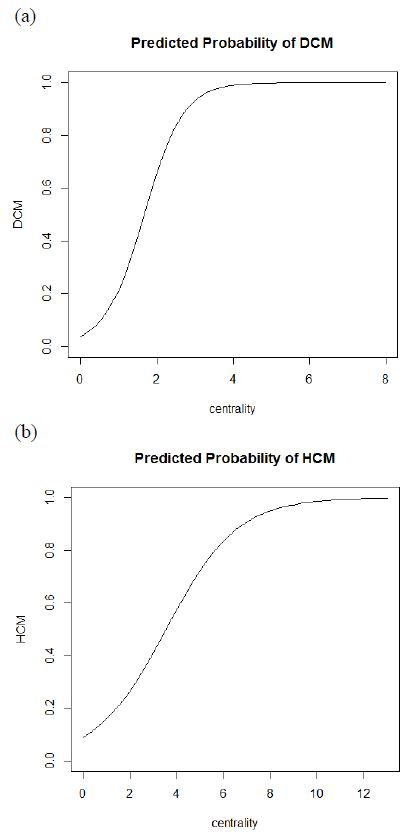
Logistic regression curve for gene selection. (a) The independent variable of the regression curve is the centrality of the causative gene of DCM and the dependent variable is the OMIM result. p-value = 8.47x10-4. (b) The independent variable of the regression curve is the centrality of the causative gene of HCM and the dependent variable is the OMIM result. p-value = 1.95x10-2.
다음으로 receiver operating characteristic (ROC)커브를 통해 회귀 모델을 평가하였다. ‘RNA-Seq identifies novel myocardial gene expression signatures of heart failure’의 연구 결과[27] 중 심부전에서 발현량이 변화하는 129개의 유전자에 대한 DCM과 HCM의 선천적인 원인이 되는 유전자들의 중심성 분석을 수행하였다. 단백질간의 interaction을 보정한 데이터를 독립변수로, OMIM데이터 베이스의 임상적 결과를 종속변수로 하는 데이터셋을 앞서 만든 회귀분석 모델에 적용하여 ROC커브의 area under the curve (AUC)를 측정하였다. AUC는 DCM과 HCM에서 각각 0.840과 0.787를 나타내 각각 good, fair한 예측정확도를 보였다(Figure 4).
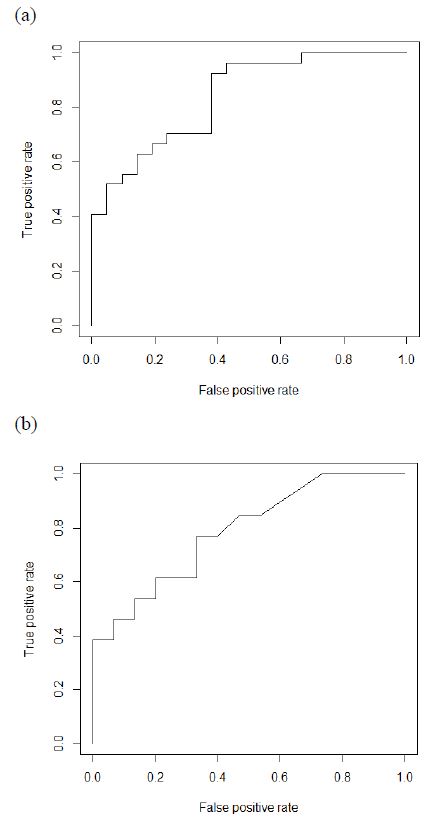
ROC curve for logistic regression curve. Centrality analysis is performed between the genes whose expression levels change in heart failure in other study and the genes responsible for DCM and HCM. The result is applied to the regression model to draw the ROC curve and calculate AUC. (a) DCM, AUC = 0.840. (b) HCM, AUC = 0.787.
위의 로지스틱 회귀 분석에서 질병의 위험도를 나타내는 적합값은 0.5 미만의 경우에는 0으로, 0.5이상인 경우에는 1로 반올림을 하여 심부전의 예후를 나타낼 것을 예측하는데, 이를 통해 얻어진 유전자들은, DCM에서는 ACTC1, ACTN2, BAG3, DES, DMD, DSG2, DSP, FKTN, ILK, LAMP2, LDB3, LMNA, MYH7, MYPN, PKD1, PKP2, PLN, PSEN1, RBM20, RYR2, SCN5A, SGCD, TCAP, TPM1, TTN, VCL이고, HCM에서는 ACTC1, ACTN2, ANKRD1, LAMP2, MYOZ2, MYPN, PLN, PRKAG2, TTN, VCL이다.
Discussion
심부전은 말기의 경우 사망률이 높고 고비용의 치료가 필요한 만큼 조기에 진단을 하고 적절한 치료를 하는 것이 심부전 환자들의 수명을 연장 시키고 삶의 질을 향상 시키는데 중요하다. 유전적인 원인에 의한 심부전의 경우 많은 경우가 심근증으로부터 비롯된다. 따라서 심근증 환자들의 예후로 심부전이 나타날 것을 예측하는 것은 적절한 치료와 모니터링을 통해 질환이 심부전으로 악화되는 것을 줄이는데 도움이 될 수 있다. 또한 의료 기술의 발전으로 Clustered regularly interspaced short palindromic repeats (CRISPR) 같이 유전자에 대한 직접적인 처치가 가능해 지고 있기 때문에 질병에 대한 분자수준의 연구는 질병의 발병 메커니즘을 이해하는데 뿐만 아니라 분자수준의 치료를 하는데 많은 정보를 제공 할 수 있을 것이다[28].
본 연구에서는 증가하는 심근증 및 심부전에 대한 유전학적 정보와 여러 생명정보학적 기법을 통해 심근증에서 심부전의 예후를 나타내는데 중요한 역할을 하는 유전자를 찾고, 순환혈액으로 이루어지는 통상적인 진단검사를 통해 심부전의 예후를 예측 할 수 있는 마커유전자를 찾고자 하였다.
Co-EGs의 1-mode 네트워크 분석에서는 HNMT, PTN, ITIH5, SMOC2, SULF1, NAMPT, FAM188A, ECM2, OIP5-AS1, C1QTNF7의 중심성이 높게 나왔는데, 이는 심근증에서 심부전의 예후가 나타나게 하는데 중요한 역할을 하는 유전자들이라 볼 수 있고 이에 대한 연구 및 치료의 타겟이 될 수 있다. DCM 및 HCM의 원인 유전자와 Co-EGs간의 2-mode 네트워크 분석에서 DCM의 경우 ACTC1, ACTN2, BAG3, DES, DMD, DSG2, DSP, FKTN, ILK, LAMP2, LDB3, LMNA, MYH7, MYPN, PKD1, PKP2, PLN, PSEN1, RBM20, RYR2, SCN5A, SGCD, TCAP, TPM1, TTN, VCL의 중심성이, HCM의 경우에는 ACTC1, ACTN2, ANKRD1, LAMP2, MYOZ2, MYPN, PLN, PRKAG2, TTN, VCL의 중심성이 각각 높게 나왔다. 이를 통해 해당 유전자들의 선천적인 변이로 인한 심근증의 경우 그 예후로 심부전이 나타날 가능성이 높다는 것을 유추 할 수 있으다. 또한 유전적인 요인으로 인한 심근증의 경우, 심부전의 예후를 예측하는데 있어 이 유전자들을 바이오마커로 활용 할 수 있을 가능성을 제시해 준다.
위에서 선별된 유전자 외에 중심성은 낮지만 OMIM 데이터베이스에서는 심부전과 관련이 있다고 나오는 유전자들은 DCM에서는 MYBPC3, TNNC1, TNNI3, TNNT2가, HCM 에서는 MYBPC3, MYH7, MYL3, TPM1이 있는데 위의 유전자들은 Kyoto Encyclopedia of Genes and Genomes (KEGG)데이터베이스의 Cardiac muscle contraction pathway에서 Calcium signaling pathway와 myosin 사이에 위치하거나 myosin과 직접 연결되는 단백질을 코딩하고 있는 유전자들이다. 이는 유전자 발현상관성과 단백질간의 interaction이 낮아도 심장의 수축/이완 과정에 중요한 역할을 하는 유전자의 변이는 심부전을 불러 올 수 있다는 것을 보여주는 결과로 사료된다.
Conclusion
본 연구에서는 선천적인 변이가 심근증의 원인이 되는 유전자들과 심근증에 대한 마이크로어레이 데이터를 이용한 소셜네트워크 분석으로 심근증의 예후로 심부전이 나타날 것을 예측 할 수 있는 유전자들을 선별하였다. 순환혈액으로 이루어지는 통상적인 진단검사로는 유전자의 선천적인 변이 외에 체세포변이나 지엽적인 유전자 발현량의 변화를 알아내기 어렵다. 그러나 여러 생명정보학적 기법을 통해 심부전이 나타나게 하는 유전자 발현 변화에 큰 영향을 끼치는 유전자들을 선별할 수 있고, 이러한 유전자들을 진단검사에 적용해 심근증의 예후로 심부전이 나타날 것 이라는 예측이 가능하게 할 것이다. 이는 심부전을 조기에 발견하여 환자들이 적절한 치료와 모니터링을 받을 수 있는 기회를 제공하는 한편, 생명정보학적 기법을 통해 마이크로어레이 데이터를 순환혈액으로 이루어지는 통상적인 진단검사에 적용할 수 있는 가능성을 보여주는 것이다.
Acknowledgement
이 연구는 2017년도 교육부와 2016년도 미래창조과학부의 재원으로 한국연구재단의 지원으로 수행되었으며(No. 2017R1D1A1B03033413 & No. 2016R1C1B2015511), 본 논문은 부분적으로 CMB의 지원을 받아 수행된 연구의 결과물 임.
References
- Elliott, P, Andersson, B, Arbustini, E, Bilinska, Z, Cecchi, F, Charron, P, Dubourg, O, Kuhl, U, Maisch, B, McKenna, WJ, Monserrat, L, Classification of the cardiomyopathies: a position statement from the European Society Of Cardiology Working Group on Myocardial and Pericardial Diseases, European heart journal, (2007).
-
McNally, EM, Barefield, DY, Puckelwartz, MJ, The genetic landscape of cardiomyopathy and its role in heart failure, Cell metabolism, (2015), 21(2), p174-182.
[https://doi.org/10.1016/j.cmet.2015.01.013]

-
Maron, BJ, Carney, KP, Lever, HM, Lewis, JF, Barac, I, Casey, SA, Sherrid, MV, Relationship of race to sudden cardiac death in competitive athletes with hypertrophic cardiomyopathy, Journal of the American College of Cardiology, (2003), 41(6), p974-980.
[https://doi.org/10.1016/s0735-1097(02)02976-5]

- Bharucha, T, Lee, KJ, Daubeney, PE, Nugent, AW, Turner, C, Sholler, GF, Robertson, T, Robert, J, Ramsay, J, Carlin, JB, Colan, SD, Sudden death in childhood cardiomyopathy: results from a long-term national population-based study, Journal of the American College of Cardiology, (2015), 65(21), p2302-2310.
-
Kensler, RW, Shaffer, JF, Harris, SP, Binding of the N-terminal fragment C0–C2 of cardiac MyBP-C to cardiac F-actin, Journal of structural biology, (2011), 174(1), p44-51.
[https://doi.org/10.1016/j.jsb.2010.12.003]

-
Maron, BJ, Niimura, H, Casey, SA, Soper, MK, Wright, GB, Seidman, JG, Seidman, CE, Development of left ventricular hypertrophy in adults with hypertrophic cardiomyopathy caused by cardiac myosin-binding protein C gene mutations, Journal of the American College of Cardiology, (2001), 38(2), p315-321.
[https://doi.org/10.1016/s0735-1097(01)01386-9]

-
Walsh, R, Rutland, C, Thomas, R, Loughna, S, Cardiomyopathy: a systematic review of disease-causing mutations in myosin heavy chain 7 and their phenotypic manifestations, Cardiology, (2010), 115(1), p49-60.
[https://doi.org/10.1159/000252808]

- Herman, DS, Lam, L, Taylor, MR, Wang, L, Teekakirikul, P, Christodoulou, D, Conner, L, DePalma, SR, McDonough, B, Sparks, E, Teodorescu, DL, Truncations of titin causing dilated cardiomyopathy, New England Journal of Medicine, (2012), 366(7), p619-628.
- Marcus, FI, Edson, S, Towbin, JA, Genetics of arrhythmogenic right ventricular cardiomyopathy: a practical guide for physicians, Journal of the American College of Cardiology, (2013), 61(19), p1945-1948.
-
Kapoor, M, Seth, S, Rao, VR, Clinical genetic aspects of cardiomyopathies, Journal of the Practice of Cardiovascular Sciences, (2015), 1(2), p120.
[https://doi.org/10.4103/2395-5414.166325]

-
Towbin, JA, Lorts, A, Jefferies, JL, Left ventricular non-compaction cardiomyopathy, The Lancet, (2015), 386(9995), p813-825.
[https://doi.org/10.1016/s0140-6736(14)61282-4]

- Go, AS, Mozaffarian, D, Roger, VL, Benjamin, EJ, Berry, JD, Blaha, MJ, Fullerton, HJ, Heart disease and stroke statistics-2014 update, Circulation, (2014), 129(3).
-
Yang, J, Xu, WW, Hu, SJ, Heart failure: advanced development in genetics and epigenetics, BioMed research international, (2015).
[https://doi.org/10.1155/2015/352734]

-
Morita, H, Seidman, J, Seidman, CE, Genetic causes of human heart failure, The Journal of clinical investigation, (2005), 115(3), p518-526.
[https://doi.org/10.1172/jci200524351]

- Go, AS, Mozaffarian, D, Roger, VL, Benjamin, EJ, Berry, JD, Borden, WB, Franco, S, (2013), Heart disease and stroke statistics-2013 update, Circulation, (2013), 127(1).
-
Lee, JH, Lim, NK, Cho, MC, Park, HY, Epidemiology of heart failure in Korea: present and future, Korean Circulation Journal, (2016), 46(5), p658-664.
[https://doi.org/10.4070/kcj.2016.46.5.658]

-
Askoxylakis, V, Thieke, C, Pleger, ST, Most, P, Tanner, J, Lindel, K, Katus, HA, Debus, J, Bischof, M, Long-term survival of cancer patients compared to heart failure and stroke: a systematic review, BMC cancer, (2010), 10(1), p105.
[https://doi.org/10.1186/1471-2407-10-105]

- Paul, A, Schinke, M, Brown, J, Riggi, LE, Izumo, S, Bartunek, J, Tsubakihara, M, Changes in cardiac transcription profiles brought about by heart failure, In Bauer Center for Genomic Research, NCBI, Gene Expression Omnibus, (2004).
- Mulligan, MK, Mozhui, K, Prins, P, Williams, RW, GeneNetwork – A toolbox for systems genetics, In Systems Genetics, Methods in Molecular Biology in press, (2016).
- GTEx Consortium, The Genotype-Tissue Expression (GTEx) pilot analysis: Multitissue gene regulation in humans, Science, (2015), 348(6235), p648-660.
- Verducci, JS, Melfi, VF, Lin, S, Wang, Z, Roy, S, Sen, CK, (2006), Microarray analysis of gene expression: considerations in data mining and statistical treatment, Physiological genomics, 25(3), p355-363.
-
Bolstad, BM, Irizarry, RA, Åstrand, M, Speed, TP, A comparison of normalization methods for high density oligonucleotide array data based on variance and bias, Bioinformatics, (2003), 19(2), p185-193.
[https://doi.org/10.1093/bioinformatics/19.2.185]

- Verducci, JS, Melfi, VF, Lin, S, Wang, Z, Roy, S, Sen, CK, Microarray analysis of gene expression: considerations in data mining and statistical treatment, Physiological genomics, (2006), 25(3), p355-363.
-
Huang, DW, Sherman, BT, Lempicki, RA, Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources, Nature protocols, (2009), 4(1), p44-57.
[https://doi.org/10.1038/nprot.2008.211]

- Ohn, JH, Kim, J, Kim, JH, Social network analysis of gene expression data, In AMIA Annual Symposium Proceedings, American Medical Informatics Association, (2003), 58.
- Dekker, A, Conceptual distance in social network analysis, Journal of social structure, (2005), 6(3).
-
Liu, Y, Morley, M, Brandimarto, J, Hannenhalli, S, Hu, Y, Ashley, EA, Tang, WH, Moravec, CS, Margulies, KB, Cappola, TP, Li, M, RNA-Seq identifies novel myocardial gene expression signatures of heart failure, Genomics, (2015), 105(2), p83-89.
[https://doi.org/10.1016/j.ygeno.2014.12.002]

- Zhang, F, Wen, Y, Guo, X, CRISPR/Cas9 for genome editing: progress, implications and challenges, Human molecular genetics, (2014), ddu125.
